Biologie quantitative : les micropuces à ADN


Principe
Le principe d’une puce à ADN repose sur la propriété qu’a l’ARN messager ou ARNm2 à se lier spécifiquement à la séquence complémentaire d’ADN.
En déterminant la concentration en ARNm, on obtient indirectement la concentration en protéines.
La première étape consiste à fabriquer la micropuce (figure 1) : chaque puits correspond à un gène du génome considéré. L’ADN de ce gène est amplifié par une méthode d’amplification très fiable nommée PCR3, puis déposé sur la surface précédemment recouverte de poly-lysine. Lorsque la goutte concentrée en ADN sèche, l’ADN adhère à la surface. Chaque puce peut contenir des milliers de puits.
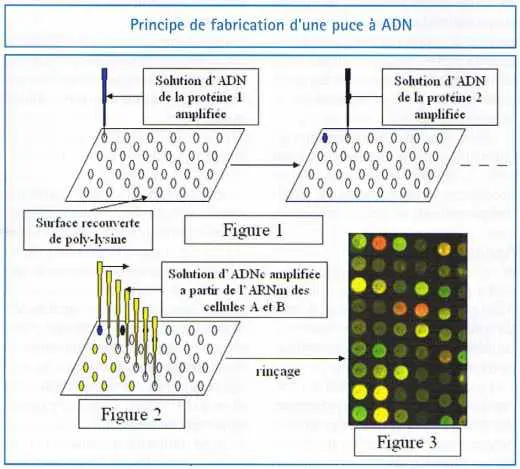
La seconde étape consiste à préparer le génome exprimé des deux cellules (A et B) à tester. Chacune des cellules à comparer est cultivée séparément, leur membrane est cassée et l’ARNm présent est amplifié par PCR sous forme d’ADN (figure 2). À cette étape, l’ADN de la population de cellules A et B est lié à un marqueur fluorescent de couleur verte et rouge respectivement.
La troisième étape consiste à comparer les niveaux d’expression de deux populations de cellules (A et B) sur la puce. L’ADN amplifié et marqué des deux cellules est mélangé. Il est ensuite piégé dans les puits par l’ADN qui lui correspond. Après un rinçage de la surface, les résultats sont observables (figure 3). Il s’agit toujours de niveaux d’expression relatifs entre les populations A et B. Les puits marqués en rouge correspondront aux gènes surexprimés dans la population B par rapport à A, ceux en verts aux gènes sous-exprimés. Les puits jaunes mettent quant à eux en évidence les gènes dont le niveau d’expression est le même dans les deux populations.
Ainsi, une puce divisée en minuscules puits sur chacun desquels serait collée la séquence codant pour chaque protéine permet d’observer le profil d’expression de génomes entiers.
Applications
La technique des micropuces à ADN permet de comparer les profils d’expression de deux populations de cellules. Trois catégories d’applications retiennent l’attention.
La comparaison des profils d’expression entre des cellules présentant des phénotypes4 différents permet de découvrir les bases moléculaires de ces phénotypes. La comparaison de tissus sains et malades permet d’identifier les gènes dérégulés lors d’une maladie. Cette stratégie est largement utilisée par les laboratoires pharmaceutiques dans leur quête de nouveaux médicaments.
La comparaison des profils d’expression au cours du temps est une source importante d’information pour comprendre des phénomènes comme la division cellulaire (Chu et al, Science 282 699).
Une analyse des profils d’expression et de leurs corrélations permet aussi de découvrir de nouvelles interactions entre protéines. Ainsi, des gènes étant exprimés simultanément ou consécutivement dans différents contextes cellulaires ont une forte probabilité d’être fonctionnellement reliés et peuvent être régulés par les mêmes éléments biologiques (clustering de gènes).
Problèmes
Les problèmes inhérents à cette technologie sont de trois ordres :
- la concentration d’ARNm n’est pas nécessairement représentative de l’activité de la protéine correspondante,
- la fabrication d’une puce à ADN est un processus cher et nécessite un matériel technologiquement avancé, donc très cher,
- le génome entier de l’organisme étudié doit être complètement séquencé et la fonction des gènes suffisamment connue (homme, souris, drosophile).
Futur
Pour observer directement et à grande échelle la présence de protéines et leur activité, les chercheurs en protéomique cherchent à développer des puces à protéines ou même des puces à cellule.
Les puces à protéines cherchent à déterminer directement et non par le biais de l’ARNm la concentration en protéines et leur conformation spatiale (qui peut correspondre à leurs degrés d’activité). Celles-ci existent sous trois formes :
- les protéines sont collées sur une surface, et seules les protéines interagissant physiquement avec celles collées à la surface seront piégées dans le puits,
- certaines protéines ont la propriété de se lier spécifiquement avec des petites molécules en fonction de leur conformation : les petites molécules fixées sur les puits piègent la protéine dont la concentration est alors observable,
- des anticorps produits pour reconnaître tout ou partie des protéines de la cellule sont fixés sur les puits et piègent la protéine dont la concentration est alors observable.
Ainsi, il est théoriquement possible de comparer à grande échelle les concentrations de différentes protéines, et de séparer les formes actives et inactives de ces protéines. Le principal obstacle de cette technologie se situe dans la découverte des petites molécules spécifiques ou dans la production d’anticorps dirigés contre chaque protéine de la cellule.
Dans le cas des micropuces à cellule, on introduit une micropuce à ADN dans un milieu contenant des cellules. Les cellules vont se multiplier dans chaque puits et vont « ingérer » l’ADN contenu (transfection). Cet ADN va induire la production de la protéine correspondante et on pourra observer l’effet de celui-ci sur les caractéristiques et le devenir de cette cellule.
Une seule expérience a été effectuée dans ce domaine (Ziauddin, J & Sabatini DM (2001) Nature 411, 107).
________________________________
1. Acide désoxyribonucléique : molécule de l’hérédité.
2. ARNm : molécule intermédiaire entre un gène codé par l’ADN et la protéine correspondante.
3. PCR ou polymerisation chain reaction : méthode d’amplification des molécules d’ADN.
4. Phénotype : apparence de la cellule.