Modèles mathématiques d’épidémies les plus élaborés : pourquoi leurs prévisions initiales sont-elles souvent excessivement pessimistes ?

Les modèles mathématiques des pandémies, y compris ceux considérés comme les meilleurs, tel celui de l’Imperial College de Londres, ont souvent fourni des prévisions initiales exagérément pessimistes. L’article donne des raisons possibles de ces erreurs et suggère quelques actions qu’il conviendrait de mener pour sortir de cette situation.
« Up to now, the effects of social distancing have predominantly been studied from a viewpoint of centrally controlled action. We argue that it is of equal importance to consider the self-initiated reactions of individuals in the presence of a contagious disease. »
Sebastian Funk, Erez Gilad, Chris Watkins, and Vincent A. A. Jansen in Proceedings of the National Academy of Sciences of the USA.
Repères
Le premier essai de modélisation mathématique du développement d’une épidémie date de 1760 (Bernouilli pour la variole). Le début du XXe siècle a vu les tentatives d’Hamer pour la peste (1906) et de Ross pour la malaria (1911). Enfin est apparu en 1927 le modèle dit compartimental de Kermack (biochimiste) et McKendrick (médecin militaire) connu par son acronyme SIR. La plupart des modèles actuels en sont issus.
Le modèle SIR : un problème traditionnel de l’antique certificat d’études primaires
Un des problèmes posés traditionnellement en fin d’études primaires aux élèves qui n’entraient pas en 6e était celui du calcul de niveau d’eau dans un réservoir alimenté par un robinet et vidé simultanément à travers une bonde. Dans le cas du modèle SIR, on a trois réservoirs : le premier, contenant initialement tout le liquide (= la population saine) et remplissant progressivement les réservoirs de niveau inférieur (« Infectés » et « Rétablis et décédés ») à travers un système de bondes et de robinets.
Cependant, une différence de taille : dans l’épreuve de feu le CEP, les débits des robinets et des bondes étaient constants, alors que dans le modèle SIR les débits sont variables et dépendent des niveaux dans les réservoirs.
Pour bien saisir les raisons pour lesquelles le SIR et les modèles dérivés donnent des résultats souvent éloignés de la réalité du développement d’épidémies, il convient de porter attention à la définition précise des paramètres régissant leur fonctionnement, traditionnellement nommés β et γ.
On considère une population N (par exemple celle d’un pays ou d’une ville) où les individus peuvent être infectés par un virus (ou une bactérie, ou encore un parasite). Cette population est divisée en 3 « compartiments » :
- S ou Sains Susceptibles d’être infectés (nombre pratiquement égal à N en début d’épidémie) ;
- I ou Infectés (quelques individus contaminés en début d’épidémie) ;
- R ou Rétablis (individus contaminés puis guéris – et, dans ce dernier cas, considérés systématiquement comme immunisés – ou décédés ; réservoir vide en début d’épidémie).
Chaque individu infecté (automatiquement considéré comme immédiatement contagieux dans ce modèle très simple) a par unité de temps en moyenne κ contacts pouvant théoriquement transmettre l’infection à d’autres membres de la population, donc κ.S/N avec des individus susceptibles d’être infectés.
Si on appelle τ la transmissibilité du virus, les I personnes infectées contaminent donc I.κ.τ.S/N individus par unité de temps, qui de ce fait quittent le compartiment S. Si on pose β = κ.τ /N on a :
dS/dt = – β S I
S’il y a I infectés pour une durée moyenne D, alors par unité de temps I/D individus migrent du compartiment I vers R par guérison ou décès. On pose habituellement γ = 1/D
Donc
dR/dt = γ I (Nous reviendrons plus loin sur cette équation « foireuse ».)
Dans ce modèle simple, si la durée de l’épidémie n’est pas trop longue, on ne tient pas compte des nouvelles naissances. Donc I = N – S – R et par voie de conséquence dI/dt = – dS/dt – dR/dt.
Ce qui conduit à :
dI/dt = β S I – γ I
Point important et qui sera discuté plus loin : on suppose dans le modèle SIR de base que, entre événements changeant radicalement la propagation de l’épidémie (confinement obligatoire, apparition d’un vaccin ou d’un traitement efficaces…) qui obligent à faire de nouveaux calculs, β et γ sont constants pendant une phase d’épidémie donnée (et différents de ceux d’une épidémie qui serait causée par un autre virus).
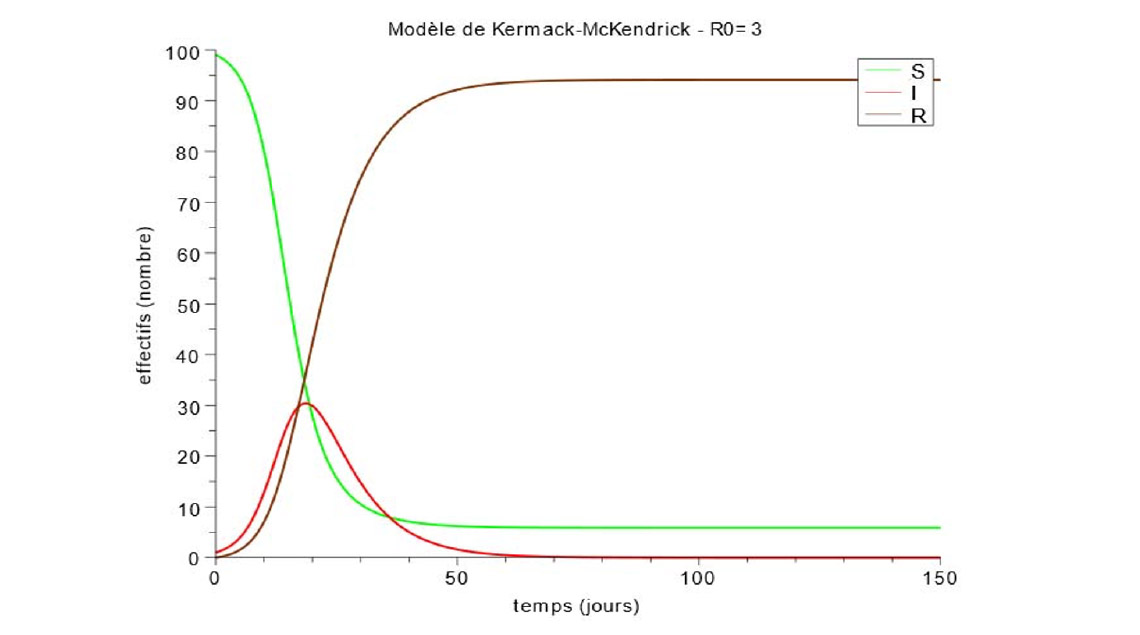
Le très médiatisé taux de reproduction initial R0 (nombre moyen de cas secondaires produits pendant toute la durée de son infection par un infectieux placé dans une population entièrement saine) est égal à κτD = βN/ γ. En début d’épidémie S est très voisin de N et la 3e équation peut alors s’écrire dI/dt = β.N.I – γ.I = γ.(R0 ‑1).I, équation de forme dI/dt = K.I ayant pour solution I = A.eKt.
À condition que R0 soit supérieur à 1, l’épidémie se développe initialement à la vitesse d’une exponentielle d’exposant γ.(R0-1).t
Lorsqu’on sort de cette zone de démarrage, l’ensemble des équations différentielles, même avec β et γ constants, n’a pas de solution analytique. Il faut avoir recours à un solveur informatique pour tracer les 3 courbes S, I et R en fonction du temps. On obtient pour I une courbe en cloche dissymétrique, la montée vers Imax étant plus rapide que la redescente vers 0. Les courbes S et R ont une forme en S dont les asymptotes donnent pour S la population restée saine en fin d’épidémie et pour R la population sortie de l’infection (guérie donc immunisée, ou décédée).

Les modèles dérivés
Depuis 1927, les chercheurs ont voulu améliorer ce modèle. Les grandes tendances sont les suivantes :
- augmentation du nombre de compartiments (infectés récents pas encore contagieux, infectés contagieux avant apparition des symptômes, séparation des guéris immunisés et des décédés, segmentation par tranches d’âges, prise en compte des asymptomatiques, des naissances pendant l’épidémie, d’une éventuelle vaccination efficace…) ;
- établissement de « matrices de contacts » permettant d’évaluer les conséquences de contacts spécifiques à différents contextes (foyer, travail, école, proximité géographique, rencontres de type sportif, culturel, confessionnel …) ;
- remplacement de données fixes par des données probabilistes afin de mettre en œuvre des modèles stochastiques ;
- utilisation de « modèles agents » fondés sur l’analyse de la conduite d’individus.
Les lecteurs qui s’intéressent à cette question prendront connaissance avec intérêt de la synthèse contenue dans l’article du 2 avril 2020 de Nature : « The simulations driving the world’s response to Covid-19 ».
Les critiques des modèles
Ce qui frappe quand on découvre ce domaine est le constat d’une prise de contrôle de fait des améliorations possibles de ces modèles par les seuls mathématiciens, qui alignent souvent pendant des dizaines de pages des suites d’équations pas toujours très compréhensibles.
« Les mathématiciens alignent souvent pendant des dizaines de pages des suites d’équations pas toujours très compréhensibles. »
Témoignent de l’incompréhension entre modélisateurs et médecins réfractaires aux équations certaines déclarations de l’épidémiologiste français le plus médiatique : « La courbe en cloche (de la Covid-19) est celle typique des épidémies. […] Les épidémies commencent, accélèrent, culminent, et elles diminuent sans qu’on sache pourquoi … C’est un cycle général habituel et on voit que c’est comme ça que se comporte cette maladie. » (Interview de fin avril 2020)
« Personnellement, je ne crois pas que la modélisation mathématique prédictive soit de nature scientifique, je pense qu’il s’agit d’une prophétie moderne comme l’a été l’astrologie à un moment donné. » (article sous sa signature dans Le Point du 3 mars 2016)
« Tous les gens qui feront des modèles prédictifs sur des maladies qu’on ne connaît pas sont des fous. » (Audition devant la commission d’enquête parlementaire, juin 2020)
Le résultat est que les modélisateurs ont généralement consacré beaucoup plus d’énergie à raffiner la partie mathématique de leurs travaux qu’à améliorer la recherche de l’exhaustivité et de la qualité des données alimentant leurs modèles, attitude à la rigueur admissible si les résultats obtenus avaient été brillants, mais les récentes et très importantes erreurs des prédictions des organismes les plus réputés montrent que ce n’est pas le cas.
Dans les épidémies réelles, des équations où β est constant ne tiennent pas compte du comportement réel de la population
Égal à κ.τ /N, β peut varier au cours du temps :
- κ est le nombre moyen de contacts de chaque individu infecté avec un individu sain pouvant théoriquement déboucher sur la contamination de ce dernier. Il dépend uniquement de facteurs comportementaux (éventuelle réduction – volontaire ou contrainte – du nombre de contacts, participation ou non à des réunions de groupes d’une certaine taille, etc.).
- τ est la transmissibilité de l’infection. Il dépend à la fois de critères médicaux (contagiosité du virus) et comportementaux (rencontre dans un espace confiné ou en plein air, port ou non de masque, fréquence du lavage des mains, gestes barrières, etc.).
La contagiosité du virus peut dépendre de facteurs externes (humidité, température, éventuellement selon un rythme saisonnier). Elle peut également évoluer lors d’éventuelles mutations de ce virus lors de sa réplication.
Il semble toutefois que ces mutations spontanées du virus ne peuvent avoir un effet global aussi rapide que des changements comportementaux qui interviennent en quelques jours, à mesure que la connaissance de l’épidémie se répand et que les autorités sanitaires imposent des mesures de protection de la population.
La plupart des modélisateurs effectuent des calculs avec des β différents avant et après adoption d’une décision de type administratif, telle que la fermeture des écoles, des salles de spectacle, le confinement généralisé, etc. Mais ils ne prennent pas en compte la réalité du comportement de la population qui prend peur à mesure que les informations alarmantes sur l’épidémie se répandent et qui change progressivement ses habitudes, puis inversement baisse la garde quand le nombre d’infectés commence à diminuer. De façon surprenante, il ne semble pas y avoir eu étude d’un modèle relativement simple où β varierait de façon continue pour tenir compte de ces comportements à mesure que le temps passe.
« La plupart des modélisateurs ne prennent pas en compte la réalité du comportement de la population. »
Notons que γ peut lui aussi varier au cours du temps, mais uniquement pour des raisons médicales : nouveaux traitements réduisant la durée moyenne d’infection, éventuelles mutations du virus.
Prise en compte de β (donc également R0) variables grâce à l’utilisation d’Excel pour la modélisation
L’utilisation d’Excel pour obtenir les résultats d’une modélisation de type SIR permet d’introduire très facilement des paramètres β (donc aussi R0) variant de façon continue avec le temps. Cette possibilité, apparemment ignorée des spécialistes, est détaillé dans un autre article de ce même numéro.
L’hypothèse de β variant pour des raisons comportementales peut expliquer des différences surprenantes entre pays
Lorsque l’épidémie de la Covid-19 est sortie de Chine, on en craignait un développement particulièrement rapide dans les pays peu développés aux structures médicales, en particulier hospitalières, très insuffisantes. Or ce sont les pays occidentaux qui ont été initialement les plus touchés.
Bien entendu les pays peu développés présentent quelques avantages face à cette pandémie, car leur population est plus jeune et encore très rurale. On peut y ajouter qu’en raison de multiples infections antérieures existent peut-être vis-à-vis d’un nouveau virus des immunisations croisées de la population (rappelons qu’il ne s’agit pas d’une notion chimérique : la première « vaccination » de l’histoire a été l’inoculation du virus de la vaccine de la vache pour prémunir de la variole humaine).
Mais les avantages de la jeunesse et de la ruralité s’estompent peu à peu : dans les dernières décennies, la longévité des individus y a augmenté de façon significative et une proportion de plus en plus importante de la population vit en ville, en particulier dans des mégapoles aux infrastructures très insuffisantes.
Les membres des sociétés avancées, quant à eux, ont une culture de confiance excessive envers la médecine depuis l’arrivée des antibiotiques et la généralisation de nombreux vaccins. Ils vivent avec le sentiment qu’ils pourront presque toujours bénéficier, s’ils contractent une maladie infectieuse, d’un traitement efficace, d’un coût pour eux réduit ou même nul dans les pays où existe un bon système d’assurances sociales. Cela peut conduire à des régressions dans certaines précautions : de nos jours, en France, avant de le remplir de lait, on rince simplement à l’eau du robinet un biberon destiné à un nourrisson, alors qu’il y quelques décennies on le laissait plusieurs minutes dans de l’eau en ébullition, réflexe de parents nés avant l’arrivée des antibiotiques.
Dans les pays peu développés, en cas d’épidémie pouvant entraîner la mort ou laisser de graves séquelles, la peur des habitants est vraisemblablement décuplée par rapport à celle de pays développés : le risque de saturation du système de santé est grand, et y accéder peut avoir un coût exorbitant pour la grande majorité de la population. Il est donc possible (mais à vérifier …) que cette peur déclenche de très brutaux réflexes de prise de distance avec les malades, ce qui pourrait plus que compenser l’hygiène médiocre accompagnant la vie courante.
Les carences dans la détermination précoce de β face à un nouveau virus
Pour être en mesure de faire des prévisions fiables, il est important de disposer dès que possible d’une estimation fiable de β à partir du nombre quotidien de nouveaux infectés. Or, dans le cas de la Covid‑19 arrivée en Europe fin 2019 ou début 2020, la plupart des pays ne connaissent toujours pas début juillet 2020 le nombre d’individus réellement infectés. Il n’est bien sûr pas possible de tester régulièrement l’ensemble de la population (à 100 000 tests par jour pour 67 millions de Français, il faudrait presque 2 ans pour un seul test par personne). Mais, dès la fin de la pénurie de tests qui avait contraint à en faire bénéficier les seules urgences médicales, il aurait été judicieux d’affecter quotidiennement une petite partie des ressources à des tests sur des échantillons médicalement représentatifs de la population. Les sociétés de sondage utilisent cette méthode pour prédire à quelques pour cent près le résultat d’élections à venir. À condition de disposer de l’aide de spécialistes médicaux, ces sociétés sont certainement capables de définir la composition d’échantillons permettant de suivre l’évolution de l’infection de l’ensemble de la population pendant plusieurs semaines.
Actuellement les données les plus fiables sont les nombres de décès quotidiens dans les hôpitaux. À partir d’évaluations de la majoration permettant de tenir compte des décès hors hôpital, de la létalité de la maladie et de la durée moyenne séparant l’infection du décès, il est possible d’établir une estimation malheureusement assez grossière du nombre d’infections quotidiennes et donc de β.
Le problème posé par l’équation « foireuse » dR/dt = γ I
Cette équation est censée traduire le fait très contestable que, s’il existe à un certain moment I infectés et si la durée de cette infection avant guérison ou décès est D, alors par unité de temps I / D individus (soit γ I) migrent du compartiment I vers R.
L’équation est une bonne approximation en régime quasi établi, mais certainement pas en régime transitoire, en particulier lors des premiers décès. En effet, si la durée moyenne d’infection est de 20 jours, il suffit qu’en début d’épidémie 20 personnes entrent dans I pour qu’en sorte presque immédiatement, selon l’équation dR/dt = γ I, une de ces 20 pour aller dans R. Dans la réalité, elle ne sera pas encore guérie !
Dans une approche de modèle simple beaucoup plus naturelle, il aurait fallu considérer que sortent à l’instant t de I ceux qui y sont entrés à l’instant (t‑D), ce qui entraînerait une équation : dR(t) / dt = – dS(t‑D) / dt. Mais manifestement Kermack et McKendrick ont voulu donner à leurs équations différentielles une allure classique afin d’en déduire des conséquences par des raisonnements d’analyse mathématique pure. Il est amusant de noter que, sans doute un peu gênés d’avoir endossé sans broncher une telle équation (qu’à la suite d’un épidémiologiste très médiatique nous pourrions qualifier de « foireuse »), les modélisateurs noient le poisson pour camoufler leur long conformisme en parlant maintenant de « discrétiser le modèle de façon à pouvoir incorporer un effet mémoire, car… un individu qui vient de rentrer dans I a une probabilité faible d’en sortir dans l’immédiat », admirable novlangue pour reconnaître que si la durée de l’infection D est fixe, le nombre de sortants de I à t est le nombre de ceux qui sont entrés dans I à (t – D) !
Conclusions
Pour que les modèles épidémiologiques soient utiles, ils devraient avant tout être, plus qu’une sorte de terrain de jeux pour mathématiciens, le résultat d’une véritable coopération multidisciplinaire entre médecins, biologistes, mathématiciens, psychologues, physiciens ou ingénieurs, instituts de sondage, tout en gardant un maximum de simplicité, et, en cas de maladie inconnue, s’appuyer dès que possible sur des évaluations quotidiennes du nombre total de personnes contaminées, guéries et décédées, obtenues par des méthodes de sondage.
De cette façon pourraient être évitées des mésaventures telles que celle de l’Hôpital Nightingale de Londres (500 lits extensible à 4 000) qui a été installé en catastrophe dans un centre de congrès, à la suite des prévisions pessimistes de l’Imperial College, et qui n’a pratiquement jamais servi.



5 Commentaires
Ajouter un commentaire
Merci pour cet article lui aussi très interessant. Je vous rejoins dans l’étonnement qu’on n’ait pas su dès le départ mieux estimer la population infectée. Il y avait en effet selon les estimations de l’institut Pasteur un facteur 20 entre les données relayées quotidiennement dans les médias et la réalité des infections. Pour autant, cette estimation pose de réelles difficultés techniques : la durée de l’infection est en réalité une courbe en cloche, dépendant elle-même de la population touchée, et il faut un certain temps et un certain nombre de cas avant d’en avoir une estimation précise et représentative.
Pour fixer les idées, on a observé un doublement des infections tous les 2,5 jours environ au début de l’épidémie. Une erreur de 2,5 jours sur la durée de contamination conduit à une erreur d’un facteur 2 sur la population infectée estimée. Pour compliquer les choses, au début de l’épidémie, on a surtout accès aux patients hospitalisés, ce qui introduit un délai supplémentaire de réaction, correspondant à la période avant hospitalisation. En définitive, quand vous passez la barre des 10 premiers morts (le 7 mars), vous croyez avoir environ 1000 cas, que vous espérez tracer, étudier et confiner, vous en avez en réalité 20 000, qui vont devenir 200 000 en 10 jours, quoique vous fassiez vraiment. Bref, il est impossible de prendre des décisions à ce moment sur une base modélisée robuste. Ce n’est qu’à peine plus facile de suivre l’évolution aujourd’hui : on peut estimer à vue de nez qu’avec 10 000 cas nouveaux par jour on doit avoir 50 000 cas réels, soit 0,1% de la population. Disons 0,7% sur une semaine. Pour avoir une précision de 0,1 point (0,1%) sur cette estimation afin de suivre son évolution, il faudrait tester chaque semaine environ 1 000 000 personnes représentatives. Ce n’est pas possible. On pourrait sans doute réduire le champ d’étude, géographiquement et par classe d’âge, mais il n’est pas garanti du tout que cela soit plus efficace que de suivre l’évolution des tests positifs déclarés, et de les redresser en fonction de la structure de la population observée.
En tout état de cause, et contrairement à ce qu’on lit dans un article d’un autre contributeur de ce numéro, le taux de mortalité chez les seniors est confirmé comme très élevé et il est très probable qu’en l’absence du confinement décidé, les prévisions pessimistes des modélisateurs auraient été bien plus proches de la réalité que celles d’un épidémiologiste médiatique très bavard.
Je ne suis pas plus spécialise du domaine qu’apparemment l’auteur de l’article, mais la naïveté de sa critique des modélisateurs m’étonne : je serais bien étonné que personne n’ait jamais joué avec un béta variant continûment au cours du temps, les modélisateurs utilisent évidemment depuis longtemps des distributions de temps (d’incubation, de contagiosité etc …) plus réalistes que celle correspondant à l’équation « foireuse » (qui est une décroissance exponentielle) etc … Rejeter les modèles plus sophistiqués sous le prétexte qu’il ne s’agirait que de dizaines de pages d’équations incompréhensibles, et ne garder que le plus simple dans sa forme la plus primitive … pour ensuite critiquer le manque de réalisme des modélisateurs : tout cela ne me semble pas très sérieux. Pas plus qu’il n’est sérieux d’invoquer les déclarations tapageuses de D. Raoult, qui soit dit en passant n’est pas épidémiologiste.
Réponse à Thierry Grenet :
Merci pour vos remarques, mais j’en regrette le ton polémique.
La consultation de https://www.researchgate.net/scientific-contributions/Thierry-Grenet-2082786602 indique qu’effectivement, tout comme moi, vous n’êtes pas spécialiste du domaine, mais chercheur au CNRS en physique du solide . Personnellement, ayant eu dans le passé une expérience de modélisation dans d’autres domaines, j’ai découvert les modèles épidémiologiques pendant le long confinement du printemps (après 20 jours de Covid !).
Pour aider le lecteur à se faire une opinion, il faut s’abstenir d’utiliser des arguments d’autorité et examiner le contenu de quelques documents représentatifs de la situation de la modélisation épidémiologique.
Pour ce faire, on peut consulter ou télécharger les documents suivants :
1 – Communication fondatrice (1927) de Kermack et McKendrick à la Royal Society de Londres https://royalsocietypublishing.org/doi/10.1098/rspa.1927.0118 Voir plus bas au début du document n° 5 la mise en évidence de « l’erreur de 1927 »
2 – « Point sur la modélisation épidémiologique pour estimer l’ampleur et le devenir de l’épidémie de COVID-19 » présentée le 30 avril 2020 à l’Office parlementaire d’évaluation des choix scientifiques et technologiques par Cédric Villani (qui, point surprenant pour un scientifique rompu à la modélisation, n’a pas relevé « l’erreur de 1927 ») https://www2.assemblee-nationale.fr/15/les-delegations-comite-et-office-parlementaire/office-parlementaire-d-evaluation-des-choix-scientifiques-et-technologiques/secretariat/a‑la-une/modelisation-epidemiologique-pour-estimer-l-ampleur-et-le-devenir-de-l-epidemie-de-covid-19
3 – « Modéliser la propagation d’une épidémie » par Hugo Falconet et Antoine Jego sous la direction d’Amandine Veber (coordinatrice de la plate-forme MODCOV19 du CNRS qui centralise des contributions relatives à la modélisation) et Vincent Calvez https://docplayer.fr/56627577-Modeliser-la-propagation-d-une-epidemie.html La raison donnée pour l’abandon des modèles déterministes décrits pendant la première partie de l’étude au profit de modèles stochastiques est incompréhensible (les modèles déterministes ne fonctionneraient pas pour des petites populations ; or les exemples de calculs donnés dans la première partie de l’étude prouvent le contraire). Voir dans la suite de la note les pages et les pages de calcul utilisant les chaînes de Markov qui n’éliminent pas « l’erreur de 1927 » (la probabilité de guérison à l’instant t ne dépendrait pas de la date de début de l’infection !).
4 – Modèles épidémiologiques (cours d’Agro ParisTech) par Suzanne Touzeau. La sortie de tous les « compartiments » utilisés dans les modèles les plus complexes est déterminée par une équation incluant « l’erreur de 1927 »
5 – « Covid-19 : « Interrogations sur le modèle épidémiologique. Prise en compte de la vaccination et du « variant anglais » » par moi-même https://www.lajauneetlarouge.com/covid-19-interrogations-sur-le-modele-epidemiologique-prise-en-compte-de-la-vaccination-et-du-variant-anglais/
Revenons à l’historique.
En 1927 Kermack et McKendrick proposent de modéliser une épidémie en divisant à tout instant une population de N habitants en 3 « compartiments » S, I et R (voir l’article). Le compartiment central des infectés pas encore guéris est vu comme un réservoir rempli par un robinet et vidé à travers une bonde, les débits d’entrée et de sortie étant variables. Dans leur modèle, le débit d’entrée ne pose pas de problème, mais le débit de sortie est considéré comme proportionnel au nombre d’infectés I contenus dans ce réservoir et donc égal à I /D, D étant la durée moyenne de contagiosité. L’équation correspondante est contradictoire en régime dynamique, croissant ou décroissant, avec l’hypothèse qu’un malade guérit (ou décède ) en général après une durée d’infection contagieuse voisine de D. Cette « erreur de 1927 » a pour conséquence que le modèle de 1927 appliqué tel quel sous-estime les variations du nombre d’infectés, que ce soit à la hausse (par exemple en début d’épidémie ou en période de déconfinement) ou à la baisse (en début de confinement).
Les successeurs de Kermack et McKendrick n’ont jamais remis en cause de façon explicite les équations de 1927, qui continuent à être enseignées sans que les réserves dues à leur inadaptation aux régimes dynamiques soient mentionnées aux étudiants. Ces successeurs ont commencé par multiplier les « compartiments » (voir le cours de S. Touzeau où subsiste « l’erreur de 1927 » pour chaque compartiment) créé des « matrices de contacts » pour tenir compte de la réalité des types de contamination, défini des profils de contagiosité variable dans le temps au cours d’une infection, introduit des hétérogénéités entre contagiosités de sous-ensembles de la population, etc. Ce sont vraisemblablement des différences persistantes entre l’évolution d’épidémies réelles et les prévisions de leurs modèles qui ont incité les successeurs de Kermack et McKendrick à aller plus loin et à donner à leurs modèles un caractère stochastique (voir plus haut la note « Modéliser la propagation d’une épidémie » supervisée par un membre du CNRS dont les longs développements mathématiques ne montrent pas clairement au lecteur d’avantage déterminant à l’utilisation de ces méthodes probabilistes). Certains vont même jusqu’à l’abandon de la notion de compartiment en recourant à des « modèles agent » peu documentés.
Ces modèles sont devenus extrêmement complexes sur le plan mathématique et très difficilement compréhensibles pour d’autres que leurs auteurs, d’autant plus que chacun semble avoir sa méthode de modélisation et que les codes informatiques qu’il emploie ne sont généralement pas rendus publics.
Les événements de 2020 (prévision pendant l’été d’une deuxième vague d’automne de montée lente démentie par les faits, inversement sous-estimation de l’effet prévisible du couvre-feu puis du confinement de fin octobre) montrent que les modèles utilisés pour préparer les décisions des pouvoirs publics souffrent du même défaut que ceux faussant les modèles simples en raison de « l’erreur de 1927 ». Apparemment, les modélisateurs professionnels ne semblent pas avoir perçu combien cette erreur pouvait perturber la conformité d’un modèle totalement ou même partiellement compartimental au développement réel d’une épidémie.
Il serait donc intéressant d’examiner avec objectivité la pertinence de modèles plus simples, mais d’où auraient été éliminées les conséquences de « l’erreur de 1927 » (ce qui est extrêmement facile).
Bonjour et merci pour votre longue réponse à mon commentaire.
Malgré ce que vous semblez penser les modélisateurs professionnels connaissent évidemment ce que vous appelez l’erreur de 1927, et savent quand elle est gênante ou pas, selon ce que l’on veut faire. S’il le faut ils l’éliminent. On peut le faire de différentes façons. Par exemple en la multipliant, si je puis dire : supposons qu’on veut décrire la cinétique du passage I–>R d’un modèle SIR. La version basique du modèle donne dI/dt=-I/D et dR/dt=I/D, soit une décroissance de type exponentielle de I (avec une probabilité de guérison indépendante de la date d’infection, comme vous le dites). Maintenant introduisez n stades intermédiaires entre I et R, appelons les J1, J2 etc, et écrivez : dI/dt=-I/D, dJ1/dt=I/D‑J1/D”, dJ2/dt=J1/D”-J2/D” etc … jusqu’à dR/dt=Jn/D”. Ce faisant vous introduisez un retard à la guérison. L’effectif total des personnes infectées (la somme de I et de tous les Jn) ne décroit plus exponentiellement, mais selon une fonction de type sigmoïde, avec une période pendant laquelle il ne se passe pas grand chose, suivie d’une décroissance concentrée autour d’une durée bien déterminée depuis la date de contamination. De cette façon on génère très facilement une distribution de type loi gamma pour la probabilité de guérison en fonction du temps écoulé depuis la contamination (on peut jouer sur n et D”).
Je ne suis pas l’inventeur de cette astuce numérique, elle fait partie de celles couramment utilisée par les modélisateurs.
Vous m’excuserez je n’ai plus son nom en tête. Essayez de l’implémenter comme je me suis amusé à la faire, c’est très facile et pratique.
Si vous lisez par exemple des papiers de l’équipe de Cauchemez à l’Institut Pasteur, vous verrez qu’ils utilisent des distributions de temps (de guérison, décès) qui essaient d’être réalistes et de correspondre aux données médicales. Ils n’en restent évidemment pas à la version de 1927 du modèle !
Cordialement
L’équation de base de la version 1927 est dI/dt =Bêta*S*I – I/D. L’idée de diviser le compartiment I en un nombre de sous-compartiments égal à la durée moyenne en jours de l’infection est intéressante si on veut introduire des niveaux d’infectiosité dépendant de l’ancienneté de l’infection (idée très bien expliquée, comme nous l’a signalé Cédric Villani dans sa récente conférence à X‑Sursaut, par l’équipe de Samuel Alizon pour son modèle développé à l’université de Montpellier. Si vous connaissez un document expliquant aussi clairement ce que fait l’Institut Pasteur, merci de me le signaler !).
Mais dans ce cas (que j’ai testé pour D=10 jours, et avec lequel je trouve les mêmes résultats que ce que je fais plus simplement avec le tableur que j’ai décrit dans un autre article), pas besoin d’une équation bien compliquée : sortent à 24 h du sous-compartiment A, celui des personnes infectées depuis X jours, pour entrer dans celui ( B ) des personnes infectées depuis X + 1 jours celles qui étaient entrées dans A à 0h (vive les lapalissades ! ). Si on veut raffiner, on peut faire rentrer directement dans R la très petite proportion des décédés pendant qu’ils étaient dans A. Pendant la journée passée dans le sous-compartiment A, les personnes infectées ont contaminé Bêta*S*I personnes saines. Quand les infectieux sortent du dernier sous-compartiment I , ils entrent dans le compartiment R. That’s all !
Que vient faire dans cette galère l’équation « foireuse » dR/dt = I/N ? C’est elle qui donne cette inepte forme fortement dissymétrique à la courbe I dans tous les cours d’épidémiologie …
Quant aux courbes sigmoïdes, ce sont celle de R et de S (I est une courbe en cloche).